数据库挖掘与分析
一、服务内容
1. 公共数据库深度挖掘
肿瘤组学:TCGA/GEO/ICGC的突变谱、表达谱、甲基化数据整合
单细胞资源:HCA(人类细胞图谱)/Single Cell Portal细胞注释与跨数据集整合
临床数据:SEER/MIMIC-IV电子病历的预后因子挖掘
药物数据库:ChEMBL/DrugBank的靶点-化合物关联网络构建
2. 私有数据建模
临床队列数据清洗(REDCap/EPIC系统导出数据标准化)
多中心研究数据去中心化处理(联邦学习框架)
实验室自产组学数据(WES/RNA-seq)与公共数据联合分析
3. 多组学关联网络
基因组-转录组-表型数据因果推断(Mendelian Randomization)
微生物组-代谢组-宿主免疫交互网络构建
药物-靶点-副作用复杂网络分析(Cytoscape可视化)
4. AI增强型分析
自然语言处理:PubMed文献知识图谱构建(BERT实体关系抽取)
影像-组学关联:放射组学特征与基因组特征联合建模
预测模型开发:基于Transformer的生存预测算法
二、服务流程
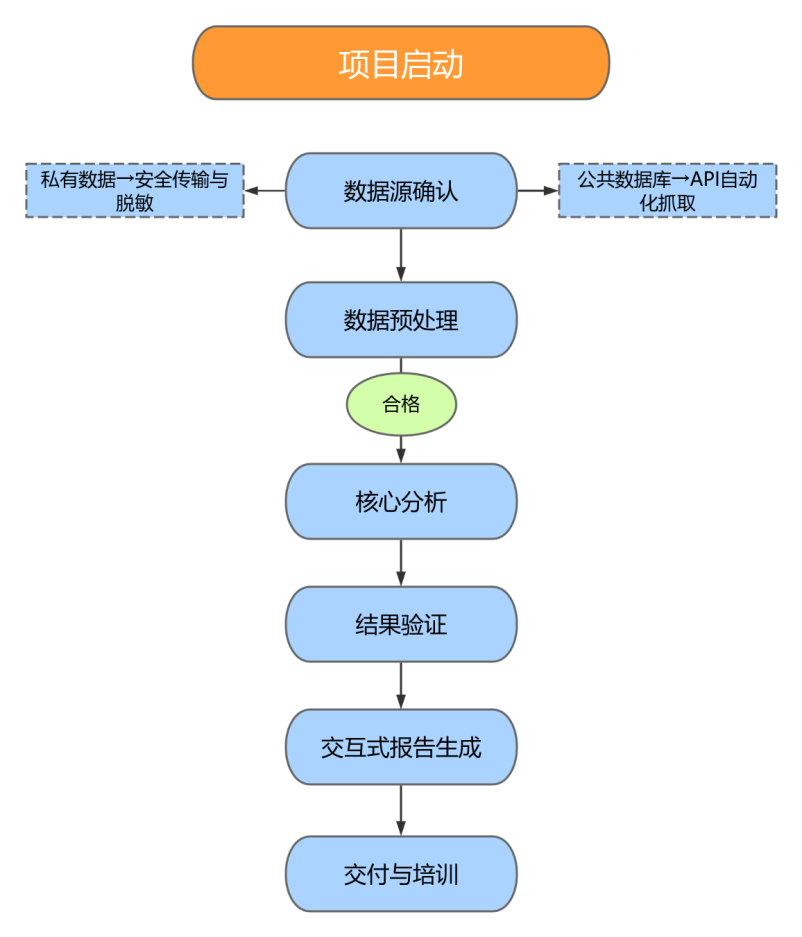
关键环节说明:
数据抓取:使用Biopython/NCBI e-utils自动化获取最新数据
预处理:
批次效应校正(ComBat/SVA)
数据归一化(TPM/RSEM/CPM标准化)
验证方法:
交叉验证(5-fold CV)
独立队列验证(如用METABRIC验证TCGA模型)
三、交付内容
类别
交付物示例
数据文件
清洗后数据矩阵(HDF5/CSV)+元数据说明文档
分析报告
PDF技术报告(含统计方法/FDR校正细节)
可视化
网络图(Gephi文件)/热图(HTML交互式)
代码
Jupyter Notebook/R Markdown可复现分析代码
四、案例示意分享
案例1:癌症生物标志物发现
数据源:TCGA乳腺癌数据集(n=1,098) + METABRIC验证队列
技术路线:
差异表达分析(DESeq2)筛选关键基因
LASSO回归构建预后模型
CIBERSORT反卷积解析免疫浸润特征
案例2:药物重定位分析
挑战:从已有药物库筛选阿尔茨海默病潜在治疗药物
方法:
构建疾病-基因-药物多层网络(基于DisGeNET/DRUGBANK)
应用deepWalk算法识别网络拓扑相似药物
分子对接验证(AutoDock Vina)
案例3:罕见病多源数据整合
需求:解析1,200例未确诊罕见病的基因组-表型关联
方案:
整合外显子数据(VCF文件)与HPO表型术语
应用Exomiser进行致病性优先级排序
构建表型相似性网络(PhenomeNET)
联系我们
地址
广州市黄埔区广州国际生物岛星汉大道31号第9层920单元
电子邮件
17620903654@163.com
联系电话
17620903654
微信
lhxs33756
微信公众号
@小童文献交流