生物信息学分析
一、服务内容
1. 基因组学分析
全基因组测序(WGS)分析:SNV/InDel/CNV/SV检测
外显子测序(WES)注释:基于ACMG指南的致病性分级
肿瘤-正常配对分析:驱动突变筛选(MutSigCV)、TMB/MSI计算
2. 转录组学分析
差异表达分析:DESeq2/edgeR流程
可变剪切分析(rMATS/SUPPA2)
lncRNA-miRNA-mRNA调控网络构建
3. 表观组学分析
ChIP-seq峰检测(MACS2)与motif富集(HOMER)
ATAC-seq开放染色质区域注释
甲基化差异分析(DSS/WGBS)与疾病风险评分
4. 单细胞多组学整合
单细胞转录组(scRNA-seq)聚类与轨迹推断(Monocle3)
CITE-seq蛋白表达与转录组关联分析
空间转录组(10x Visium)区域特异性基因表达解析
5. 宏基因组与代谢组
16S/ITS测序:物种组成分析(QIIME2)、LEfSe差异物种筛选
宏基因组功能预测(PICRUSt2/KEGG)
代谢组-微生物组联合分析(Sparse PLS-DA)
6. 数据挖掘与建模
机器学习:基于TCGA数据的生存预测模型(PyCaret)
多组学整合:WGCNA+通路富集联合分析
个性化分析流程开发(Python/R Shiny工具包)
二、服务流程
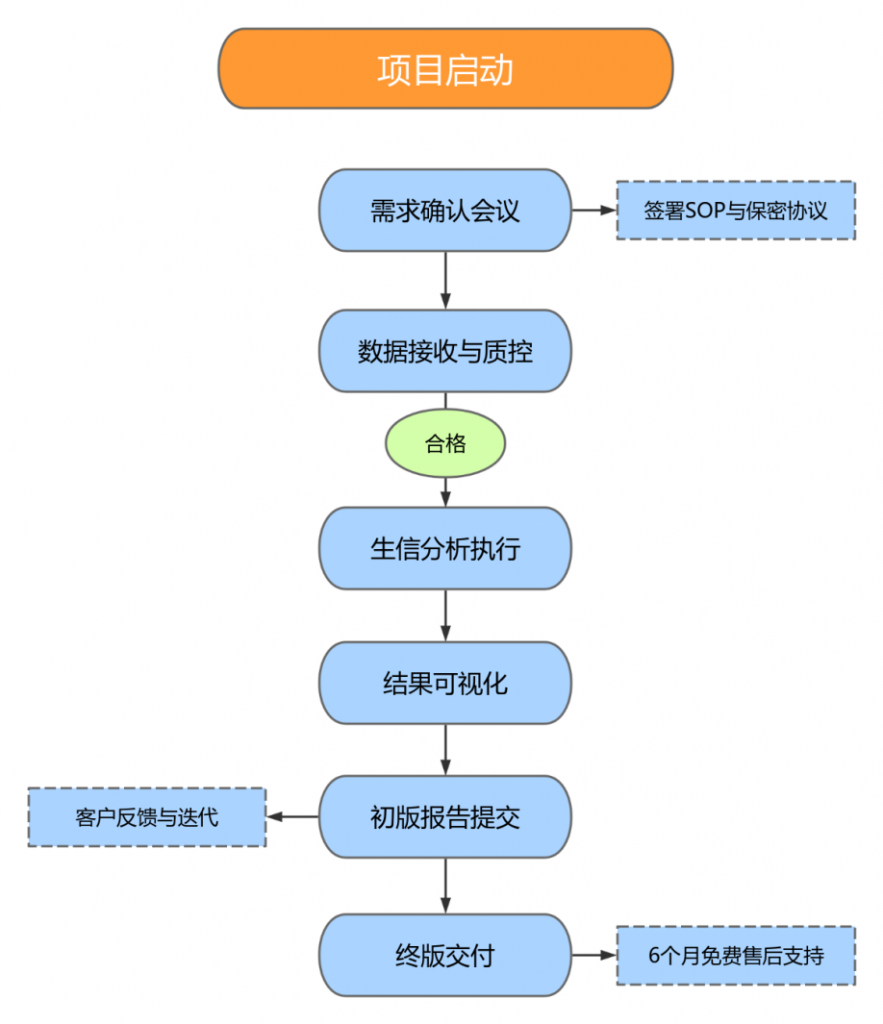
关键节点说明:
数据质控:FastQC+MultiQC报告(含测序质量/Q30/污染检测)
分析执行:使用标准化流程(GATK/GENCODE v38)
可视化:提供交互式图表(UCSC基因组浏览器/Scanpy可视化)
迭代:支持关键结果重新计算(调整差异基因阈值)
三、交付内容
类别
交付物示例
数据文件
变异注释VCF文件 / 差异基因表达矩阵(CSV)
分析报告
PDF技术报告(含方法学描述/关键图表/统计学参数)
可视化
Circos图 / 火山图 / UMAP降维图(PDF/EPS格式)
代码
分析流程脚本(Snakemake/Nextflow代码库)
高阶可选模块
交互式报告:HTML格式可点击热图/动态生存曲线
数据库支持:MySQL存储变异数据 / Neo4j构建分子互作网络
生信培训:定制化生物信息学分析实操培训(线上/线下)
四、案例示意分享
案例1:癌症基因组学
客户需求:解析肺鳞癌患者靶向治疗耐药机制
分析流程:
WES检测50对肿瘤-正常样本
使用GISTIC2.0识别显著扩增/缺失区域
整合TCGA数据筛选驱动基因(EGFR-T790M突变丰度<5%)
构建PDX模型验证NF1缺失与MEK抑制剂敏感性关联
案例2:单细胞免疫图谱
技术挑战:解析COVID-19患者外周免疫细胞动态
方案亮点:
10x Genomics单细胞转录组分析(n=8患者,>50,000细胞)
使用CellPhoneDB解析免疫细胞互作网络
Trajectory分析揭示CD8+T细胞耗竭路径
案例3:宏基因组与代谢组联合
数据规模:300例肠道样本(宏基因组+LC-MS代谢组)
创新方法:
MaAsLin2筛选疾病相关菌群-代谢物关联对
构建随机森林模型诊断结直肠癌(AUC=0.89)
体外培养验证产丁酸菌群抑制肿瘤细胞增殖
联系我们
地址
广州市黄埔区广州国际生物岛星汉大道31号第9层920单元
电子邮件
17620903654@163.com
联系电话
17620903654
微信
lhxs33756
微信公众号
@小童文献交流